|
| 1 | +# 生信爱好者周刊(第 180 期):有了AI,还有必要学习生物信息吗? |
| 2 | + |
| 3 | +这里记录值得分享的生信相关内容,每半月发布。 |
| 4 | + |
| 5 | +本杂志开源(GitHub: [openbiox/weekly](https://github.com/openbiox/weekly "openbiox/weekly")),欢迎提交 issue,投稿或推荐生信相关内容。 |
| 6 | + |
| 7 | +[「生信周刊讨论区」](https://github.com/openbiox/weekly/discussions "「生信周刊讨论区」") |
| 8 | + |
| 9 | +# 封面图 |
| 10 | + |
| 11 | + |
| 12 | + |
| 13 | +本周话题:[有了AI,还有必要学习生物信息吗?](https://mp.weixin.qq.com/s/GlMs2iRTkz9AqEBPOWG8ww) |
| 14 | + |
| 15 | +> 这篇文章通过汇集多个主流AI模型的观点,共同论证了在AI时代学习生物信息学的必要性。核心结论认为,AI不会取代生物信息学,而是作为强大的工具,改变了学习的重点:从业者需要从重复性的“代码搬运”和“跑流程”中解放出来,转而更深入地掌握生物学知识、统计学原理,并培养利用AI定义科学问题、设计分析策略及解释复杂结果的高阶能力。 |
| 16 | +# 生信研究 |
| 17 | +1、[Cell Discovery | 泛黑色素瘤的生物学和治疗的蛋白基因组学研究](https://mp.weixin.qq.com/s/WGCFQGKhFrCBqUPnhL86UQ) |
| 18 | +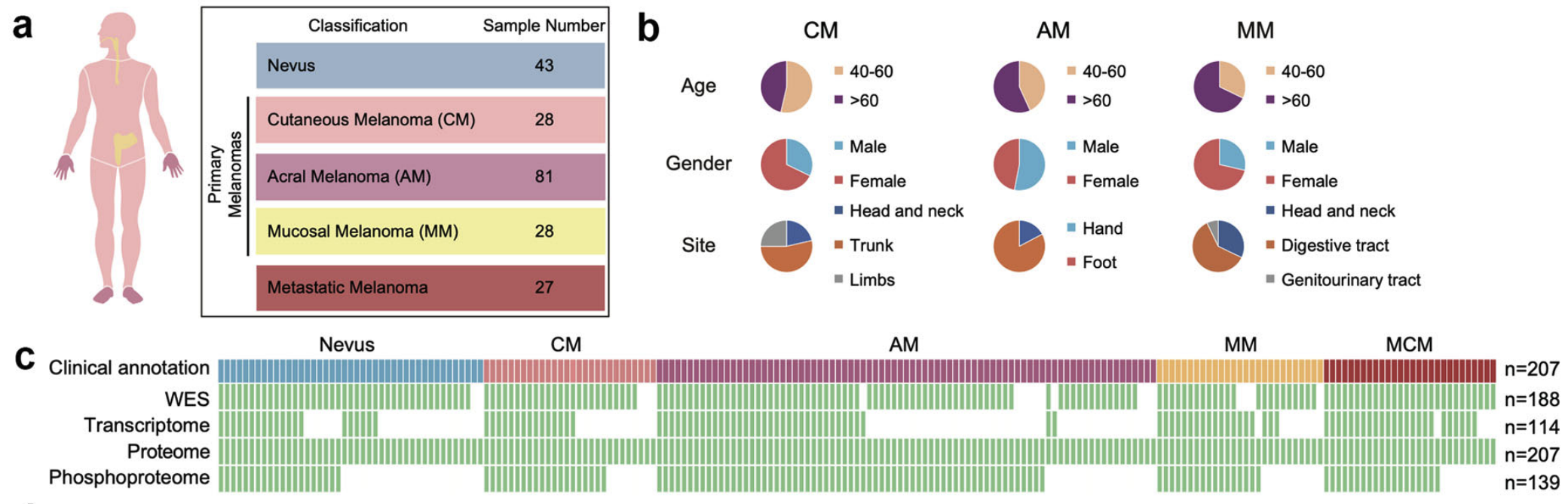 |
| 19 | + |
| 20 | +复旦大学丁琛团队等通过对包含肢端、皮肤、黏膜等亚型的207例中国黑色素瘤患者队列进行多组学整合分析,系统描绘了其蛋白基因组图谱。研究揭示了与不良预后相关的SBS7a突变特征及PRKDC基因扩增通过激活叶酸代谢促进肿瘤增殖的机制,并建立了与预后相关的蛋白质组分子分型及免疫分型,为黑色素瘤尤其是亚洲高发亚型的精准治疗提供了新的潜在靶点和分型依据。 |
| 21 | + |
| 22 | +- 论文链接:https://www.nature.com/articles/s41421-024-00688-7 |
| 23 | + |
| 24 | +2、[Nat Mach Intell | 清华大学自动化系闾海荣等发表精准病理诊断AI基础模型,可实现对胶质瘤多种分类任务的准确诊断](https://mp.weixin.qq.com/s/b1Ue3nK04hv9ZKLJ7VFv8A) |
| 25 | + |
| 26 | +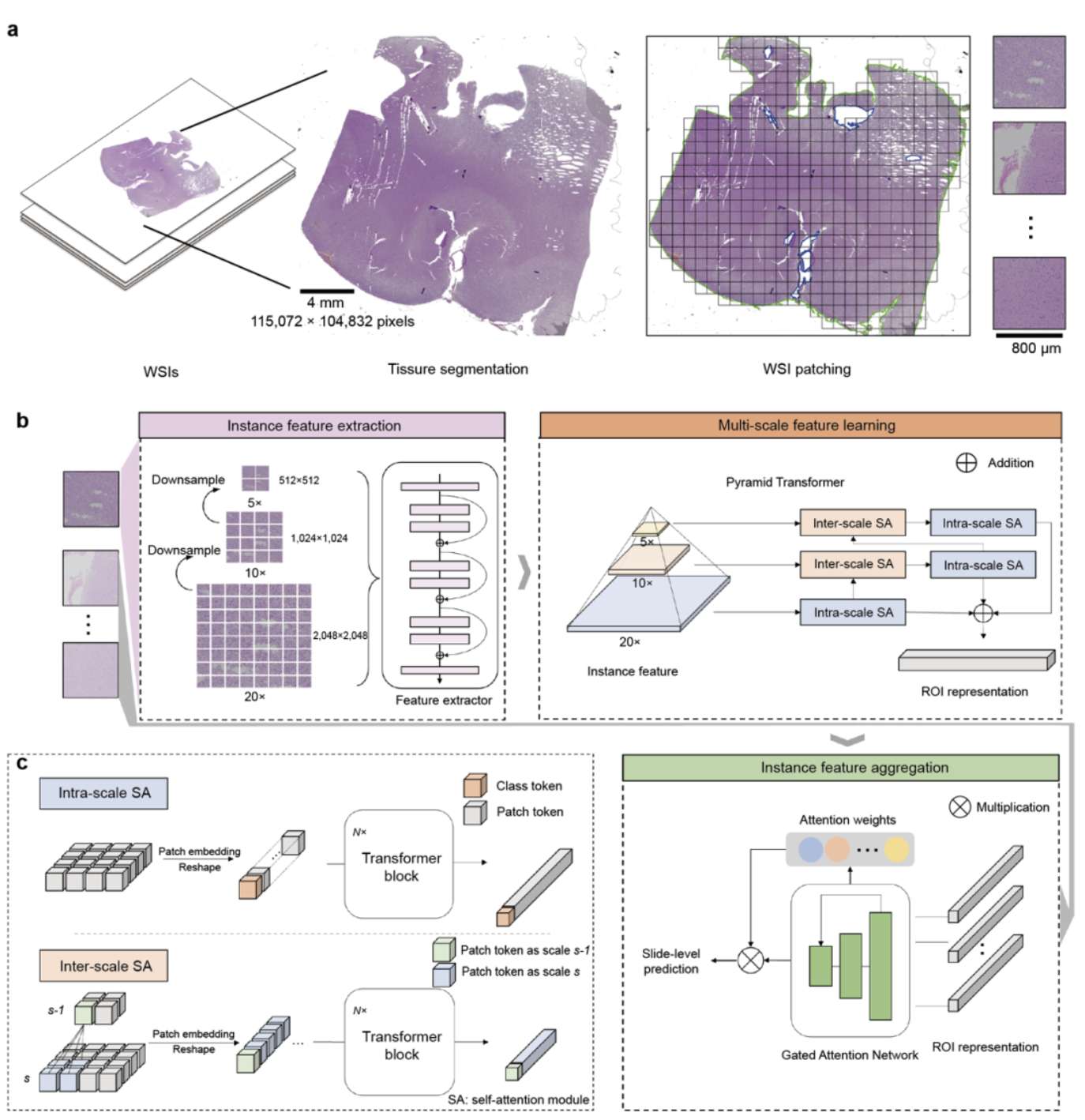 |
| 27 | + |
| 28 | +清华大学等研究团队提出了一种名为ROAM的弱监督计算病理学AI基础模型。该模型通过采用大尺寸组织图像块作为分析单元,并利用金字塔Transformer架构系统学习尺度内及尺度间的相关性特征,从而高效提取百亿像素级全切片病理图像的多尺度视觉表征。ROAM在胶质瘤的肿瘤检测、亚型分类、分级和分子特征(如IDH突变)预测等多种诊断任务上实现了临床级的高性能与良好泛化能力,其提供的可视化依据不仅能辅助病理医生提升诊断准确性,还有助于发现与分子特征相关的形态学标志物。 |
| 29 | + |
| 30 | +- 论文链接:https://www.nature.com/articles/s42256-024-00868-w |
| 31 | + |
| 32 | +3、[Nat Genet | 基于WGS数据绘制癌症进展过程中ecDNA扩增图谱,揭示抗癌新靶点](https://mp.weixin.qq.com/s/AOkdOyQtvc7jUNIuxNZrXQ) |
| 33 | + |
| 34 | +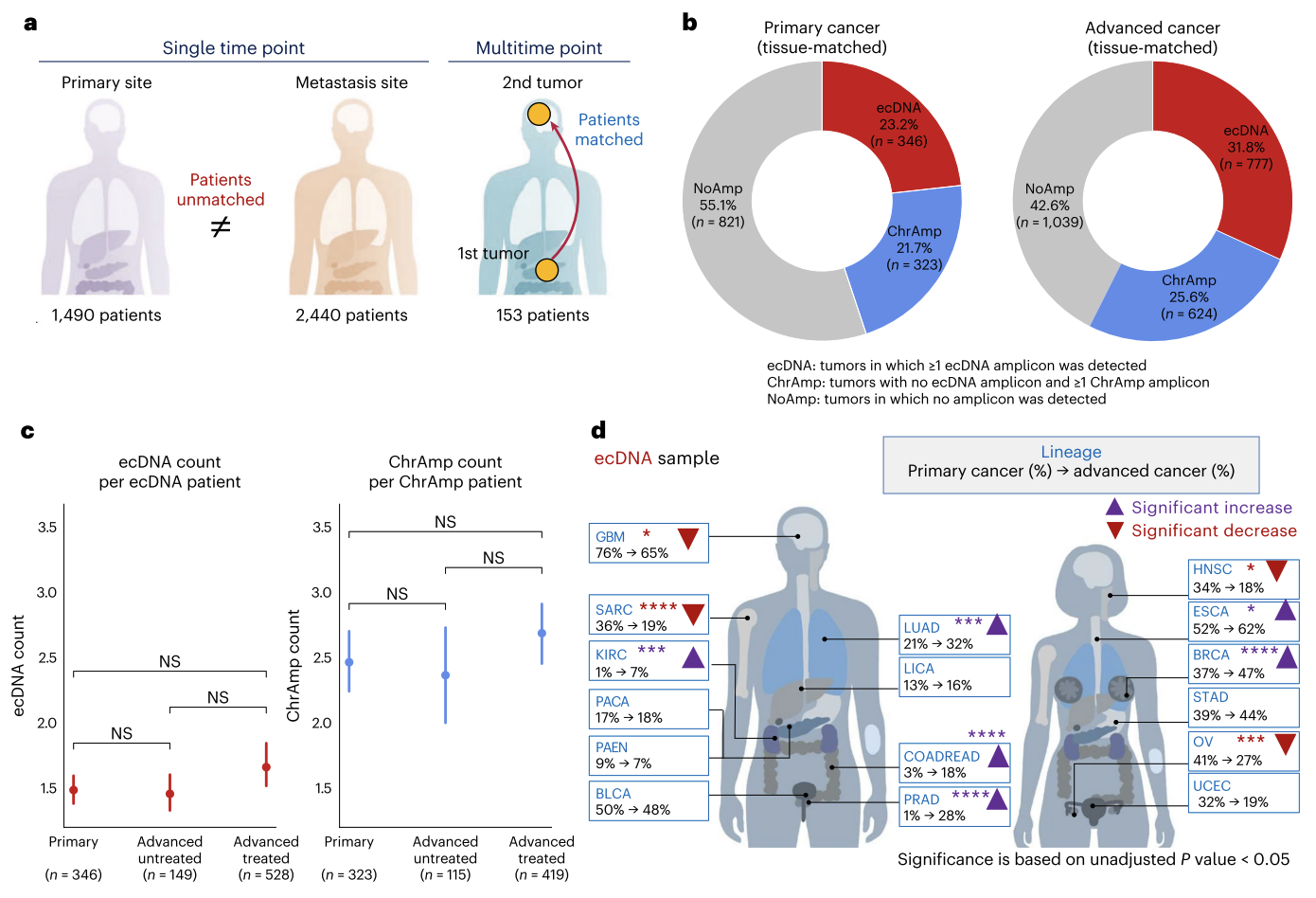 |
| 35 | + |
| 36 | +耶鲁大学医学院等研究团队通过对8060个涵盖新诊断原发性、未经治疗转移性及经治疗肿瘤的全基因组测序数据进行统一分析,系统绘制了癌症进展中染色体外DNA(ecDNA)的扩增图谱。研究发现,与早期肿瘤相比,未经治疗的转移瘤和接受过治疗的晚期肿瘤中ecDNA的出现频率显著升高,其中微管蛋白抑制剂治疗与ecDNA增加的相关性最强。研究还揭示,在肿瘤进展过程中,ecDNA相较于染色体扩增(ChrAmp)更有可能被克隆选择所保留,且其更易积累局部高突变事件。这些发现确立了ecDNA作为癌症进展的关键驱动因素和潜在治疗靶点。 |
| 37 | + |
| 38 | +- 论文链接:https://www.nature.com/articles/s41588-024-01949-7 |
| 39 | + |
| 40 | +# 博文咨询 |
| 41 | +4、[Rb的发现:揭开抑癌基因的秘密](https://mp.weixin.qq.com/s/luVxe9wgAlOHYAO3EpSO_A) |
| 42 | + |
| 43 | + |
| 44 | + |
| 45 | +20世纪70年代,克努森提出“二次突变假说”解释视网膜母细胞瘤的成因。80年代中期,麻省眼耳医院的萨德·德里亚医生通过建立肿瘤样本库并利用DNA探针技术,在肿瘤细胞中定位到第13号染色体上的关键缺失片段。随后,他与怀特黑德研究所的史蒂夫·弗兰德合作,于1986年在《自然》杂志上正式确认了该基因——即视网膜母细胞瘤易感基因(Rb)。这一发现不仅揭示了该儿童眼癌的遗传机制,更首次鉴定出一个具有广泛意义的抑癌基因,其功能失活被证明与多种成人癌症(如肺癌、乳腺癌)的发生密切相关,为理解细胞周期失控和肿瘤发生奠定了基石。 |
| 46 | + |
| 47 | +5、[读《为什么批评测序?》有感 ](https://mp.weixin.qq.com/s/M_gjrL7JmkYpxMOXvSAGwg) |
| 48 | + |
| 49 | +数据驱动的研究范式(如GWAS)作为传统假设驱动范式的必要补充,其产出的优质数据具有“长尾效应”,能随时间持续产生价值。当前真正的“浪费”在于数据共享机制的不足,导致大量数据被封闭,未能像TCGA等项目那样通过开放共享激发巨大的科研倍增效应。因此,提高数据整合与开放水平,才是释放测序数据价值、对科研资源负责的关键。 |
| 50 | + |
| 51 | +6、[利用MCP在Cursor中访问Obsidian数据进行交互 ](https://mp.weixin.qq.com/s/7XZVwPHFGSRMGc-biMsRdA) |
| 52 | + |
| 53 | + |
| 54 | + |
| 55 | +通过安装Node.js、MCP-Obsidian连接器并进行相应配置,实现了让AI助手(如Copilot)读取和搜索Obsidian笔记的功能。 |
| 56 | + |
| 57 | +# 工具 |
| 58 | +7、[Sniffnet](https://github.com/GyulyVGC/sniffnet "Sniffnet") |
| 59 | + |
| 60 | +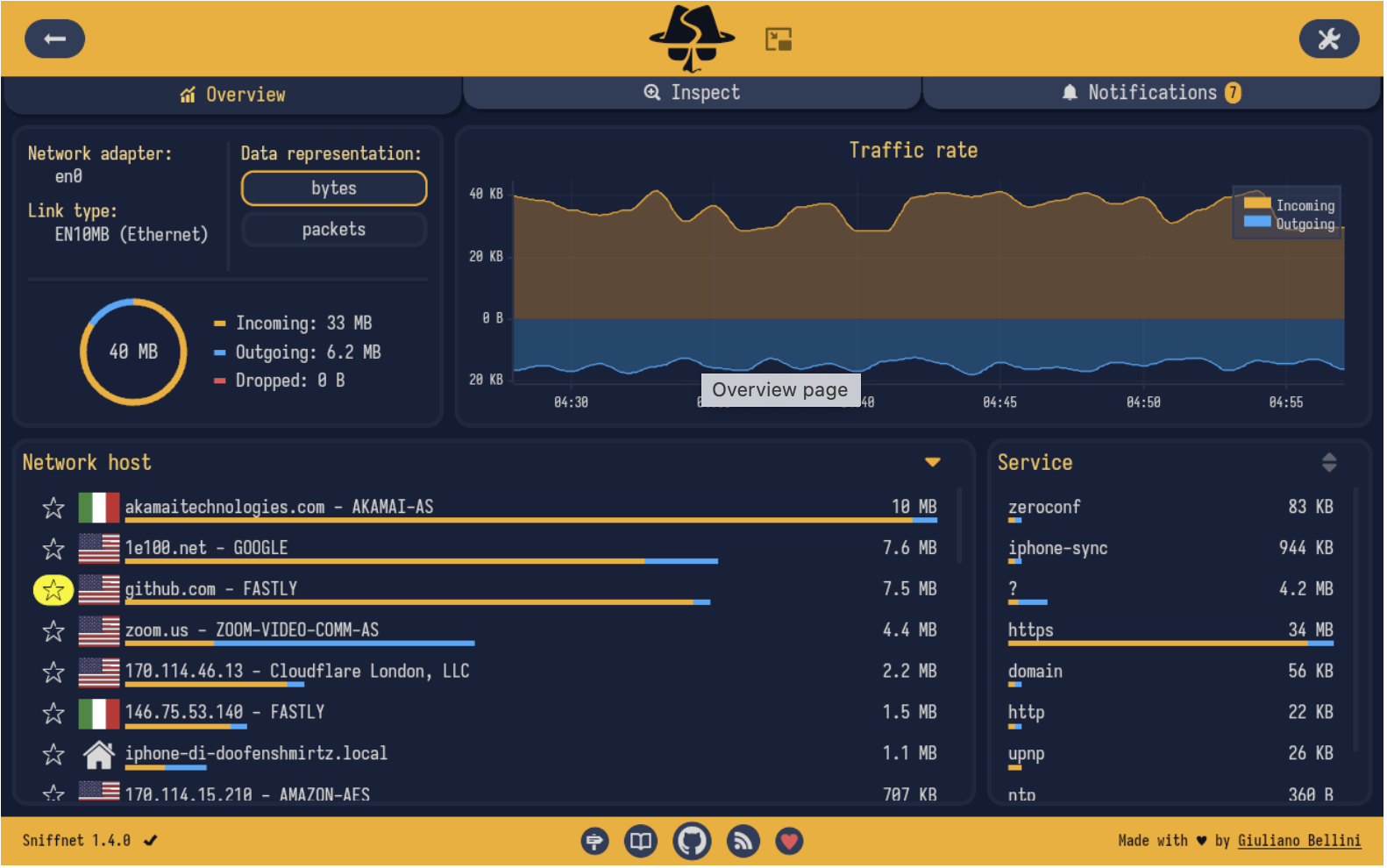 |
| 61 | + |
| 62 | +一款可以跨平台使用的、可以便捷监控网络流量的应用程序。 |
| 63 | + |
| 64 | +8、[legendry](https://github.com/teunbrand/legendry "legendry") |
| 65 | + |
| 66 | +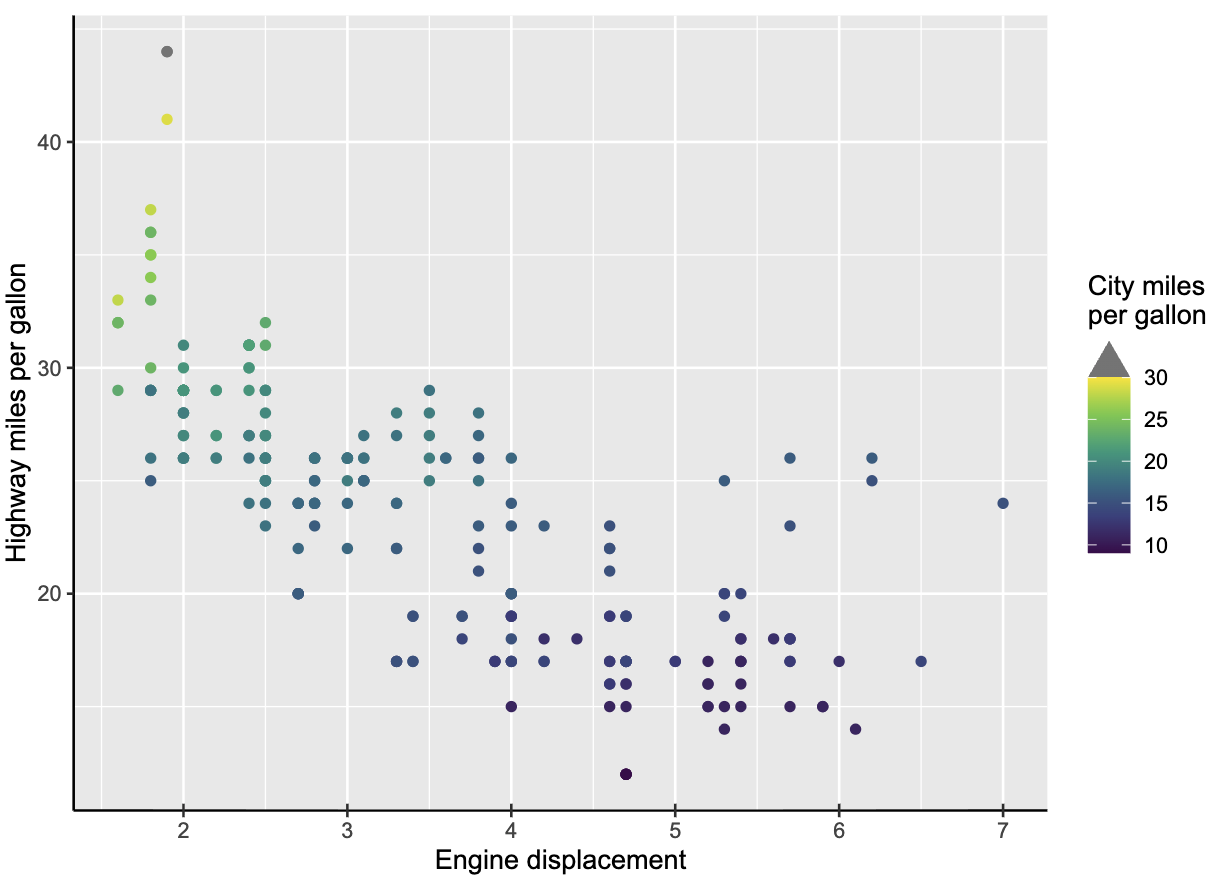 |
| 67 | + |
| 68 | +ggh4x是一个专注于扩展和增强ggplot2中“引导元素”(guides,如坐标轴、图例、颜色条)的R语言扩展包。它通过重新实现现有引导元素来提供更强大的自定义功能(如灵活调整刻度位置、格式和分面参数),并引入了全新的引导元素类型(如针对二元变量或嵌套刻度的图例),旨在帮助用户,特别是在处理具有复杂分层结构或多维度的生物信息学数据时,创建信息更丰富、展示更精确的统计图形。 |
| 69 | + |
| 70 | +9、[daily-arXiv-ai-enhanced](https://github.com/dw-dengwei/daily-arXiv-ai-enhanced "daily-arXiv-ai-enhanced") |
| 71 | + |
| 72 | +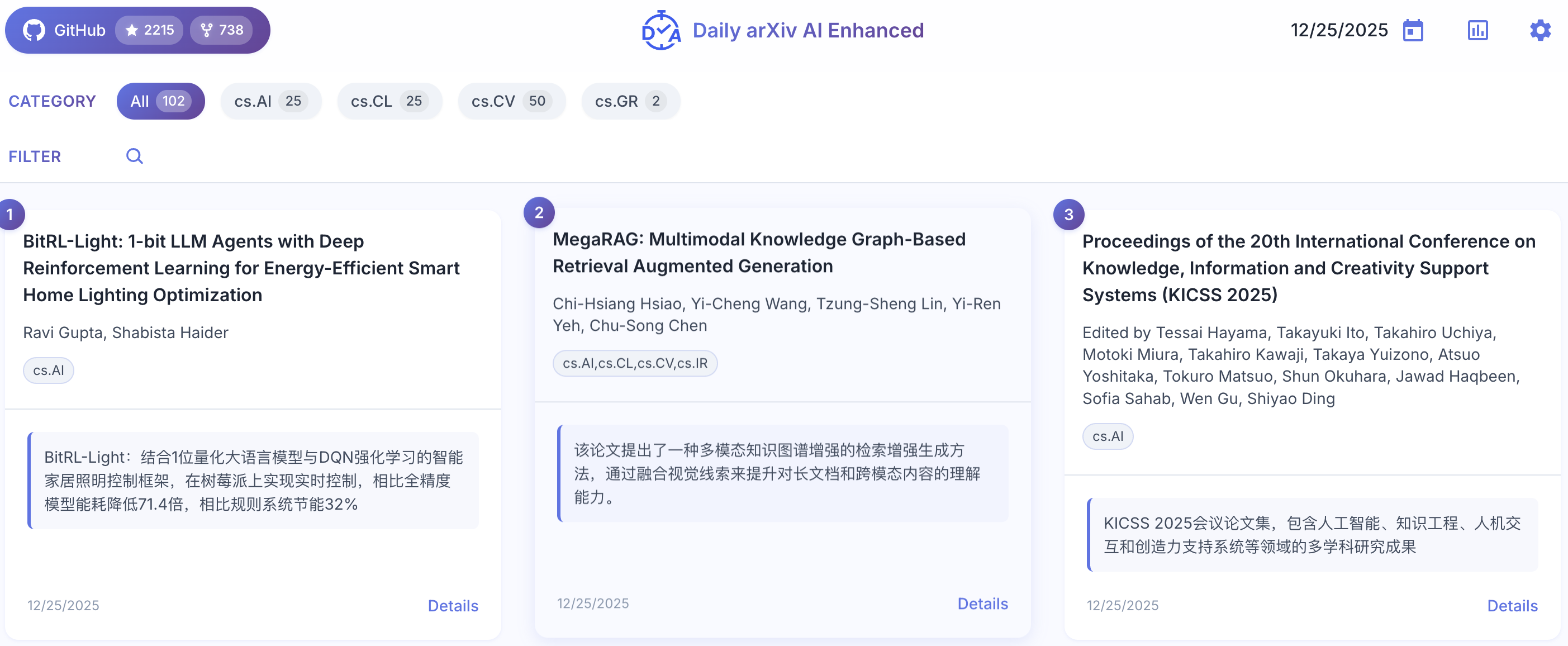 |
| 73 | + |
| 74 | +该工具实现了自动化工作流:每日抓取arXiv上的新论文,使用人工智能模型对其生成摘要,并将结果部署到GitHub Pages上完成可视化呈现。 |
| 75 | + |
| 76 | +# 资源 |
| 77 | +10、[Cell Host & Microbe | 人体肠道微生物蛋白质结构组数据库](https://blog.csdn.net/woodcorpse/article/details/155412866) |
| 78 | + |
| 79 | +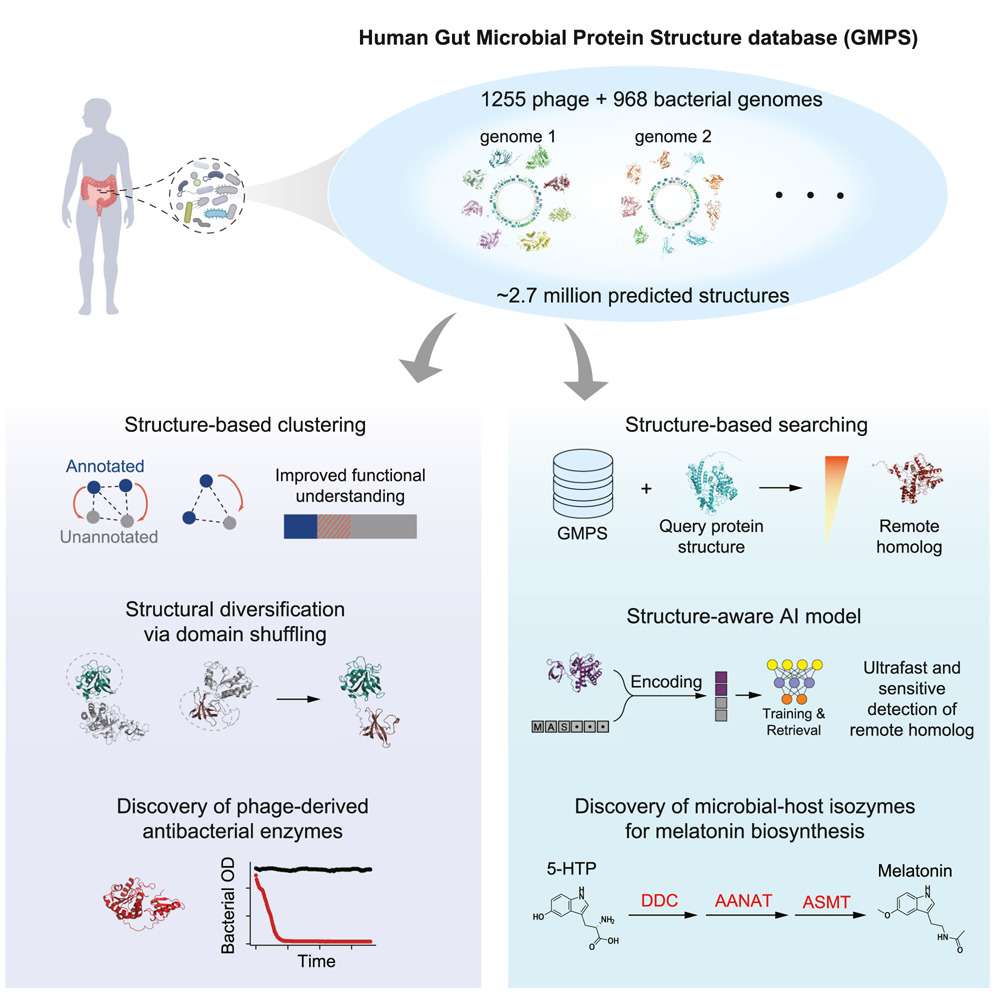 |
| 80 | + |
| 81 | +该研究建立了人体肠道微生物的蛋白质结构组数据库和结构检索方法,显著提高了对噬菌体蛋白、菌源宿主同工酶等功能暗物质的预测能力。基于这一方法,研究团队成功验证了肠道致病菌的噬菌体裂解酶,并首次揭示了肠道细菌的褪黑素合成途径。 |
| 82 | + |
| 83 | +- 论文链接:https://www.sciencedirect.com/science/article/pii/S193131282500455X#undfig1 |
| 84 | + |
| 85 | +11、[Advanced Science | 多物种单细胞AI数据库](https://cms.casdc.cn/article/721) |
| 86 | + |
| 87 | +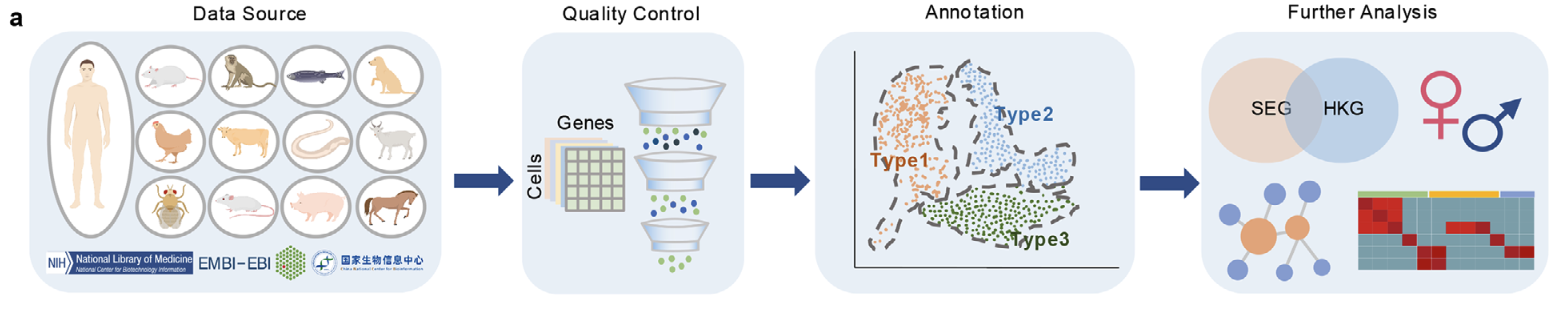 |
| 88 | + |
| 89 | +中国科学院计算机网络信息中心与动物研究所等团队联合发布了scCompass数据库。该数据库整合了覆盖人类、小鼠等13个物种、超过1亿个高质量单细胞的转录组数据,旨在为生命科学领域的大模型研发和基础生物学研究提供高标准、一站式的数据底座与服务。 |
| 90 | + |
| 91 | +- 论文链接:https://advanced.onlinelibrary.wiley.com/doi/10.1002/advs.202500870 |
| 92 | + |
| 93 | +# 贡献者(GitHub ID) |
| 94 | + |
| 95 | +「Openbiox 生信周刊」运维小队: |
| 96 | + |
| 97 | +- @ShixiangWang(王诗翔) |
| 98 | +- @kkjtmac(阚科佳) |
| 99 | +- @NiEntropy(赵启祥) |
| 100 | +- @He-Kai-fly(何凯) |
| 101 | +- @JnanZhang(张佳楠) |
| 102 | +- @Tomcxf(陈啸枫) |
| 103 | +- @wangdepin(王德品) |
| 104 | +- @kongjianyang(空间阳) |
| 105 | +- @donghongyu2020(董弘禹) |
| 106 | +- @DrRobinLuo(罗鹏) |
| 107 | +- @Wangcy-rachel(王春阳) |
| 108 | +- @zoe3251(舒晨阳) |
| 109 | +- @yanbin85 (严彬) |
| 110 | +- @MadDERt(王章宇) |
| 111 | +- @yepu03(孙裕钧) |
| 112 | + |
| 113 | +# 订阅 |
| 114 | + |
| 115 | +这个周刊每周日发布,同步更新在微信公众号「生信协作组」(elegant-r)上。 |
| 116 | + |
| 117 | +微信搜索“生信协作组”或者扫描二维码,即可订阅。 |
| 118 | + |
| 119 | + |
| 120 | +(完) |
0 commit comments